中国科学院上海有机化学研究所生物与化学交叉研究中心朱正江研究员课题组在Nature Communications杂志在线发表了题为“Metabolite Annotation from Knowns to Unknowns through Knowledge-guided Multi-layer Metabolic Networking”的研究论文 (Nature Communications, 2022, 13: 6656) 。该工作发展了一种知识驱动的多层代谢网络技术(Knowledge-guided multi-layer networking, KGMN),在复杂生物样本中实现了未知代谢物的大规模鉴定。朱正江课题组毕业生周智伟博士,博士研究生罗名都同学是论文的共同第一作者,中国科学院上海有机化学研究所生物与化学交叉研究中心为第一单位。
生命体代谢组不仅包含内源性产生的已知代谢物,还包含微生物菌群、植物、食物和其他来源的未知代谢物。这些未知代谢物对生命活动起到重要的调控作用。然而,如何大规模鉴定未知代谢物是当前代谢组学研究中的热点和难点。在前期研究中,朱正江课题组发展了基于代谢反应网络的代谢组学技术MetDNA(Nature Communications, 2019, 10: 1516)。该技术利用代谢反应网络中产物和底物存在结构相似性和二级质谱图相似性的基本原理,设计了代谢物二级质谱图“谱图借用”和“多次迭代”算法,利用标准谱图库鉴定出的代谢物作为种子,依靠代谢反应网络进行代谢物注释的迭代和传递,突破了标准二级质谱图库的覆盖度限制,实现大规模的已知代谢物鉴定。然而该技术仍然存在一些局限:1) 代谢物鉴定传递的过程是仅限于已知代谢反应网络,因此无法用于发现新的未知代谢物;2) 复杂质谱数据中的大量冗余信号 (如同位素峰、加合物峰、中性丢失和源内裂解等)会对代谢物的鉴定造成假阳性。
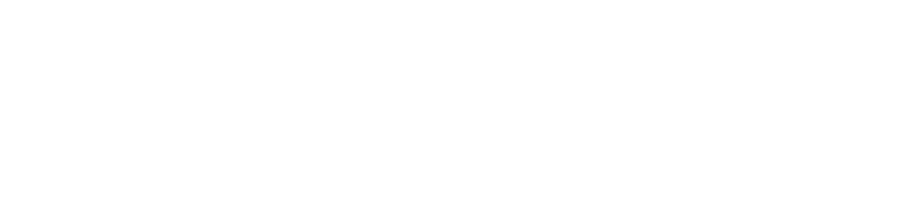
针对以上问题,作者进一步发展了知识驱动的多层代谢网络技术(KGMN),实现了从已知代谢物鉴定未知代谢物的能力,并显著提升了代谢物鉴定的准确度。基于该技术,作者开发了第二代MetDNA软件(MetDNA2)。该技术首次整合了3层代谢网络(图1):1) 知识驱动的代谢反应网络;2) 知识引导的二级质谱图相似性网络;3) 全局代谢峰相关性网络。首先,作者利用理论代谢反应对于已知的代谢反应网络进行扩展,从而构建了包含已知和未知代谢物的扩展代谢反应网络(KMRN,网络1)。MetDNA2从标准谱图库鉴定出的种子代谢物出发,基于扩展代谢反应网络和“谱图借用”策略,构建二级质谱图相似性网络(网络2)。该策略可以通过多次迭代和循环扩增的算法,将代谢组学质谱数据中的所有已知和未知代谢物连接,直到没有新的注释代谢物。在网络2中,代谢物节点之间的连接有四个限制条件:MS1 m/z、保留时间、MS/MS谱图相似性和代谢反应转化(metabolic biotransformation)。对于注释到的每一个代谢物,MetDNA2会进一步通过靶向检索其相关的冗余质谱特征峰(如同位素峰、加合物峰、中性丢失和源内裂解等),并构建全局代谢峰相关性网络(网络3)。最后,利用全局代谢峰相关性网络,MetDNA2对注释的代谢物结果进行全局优化,提升代谢物鉴定的准确度,去除假阳性注释结果返回最终鉴定结果。整个数据处理流程全程自动化,无需人工干预,提升了数据分析的效率。
图1 知识驱动的多层代谢网络技术KGMN
利用上述技术,MetDNA2对于已知代谢物的鉴定准确性从~70%提升至>95%。同时,在不同的生物样本中,MetDNA2还能够鉴定~100-300个未知代谢物;对于单个生物样本鉴定的代谢物数目在2000-5000个左右。在MetDNA2中,每一个鉴定结果均根据国际代谢组学协会标准指定特定的可信度。此外,MetDNA2还包含了一系列重要的更新和升级,如全面升级的标准代谢物谱图数据库(>2000个代谢物);两种不同色谱体系的保留时间数据库 (HILIC 和C18体系);适配所有厂商的高分辨二级质谱数据等。
为了方便相关领域研究者应用该工具,课题组提供了用户友好型的界面和网站MetDNA2 (
http://metdna.zhulab.cn/),学术用户可以免费注册使用。该工作所开发的KGMN技术已经申请了国家发明专利和国家软件著作权。相关技术和软件的商业用途需要联系朱正江研究员进行授权使用。该工作得到了国家自然科学基金委、科技部、中国科学院、上海市科委等的资助。