上海有机所在阐述新颖黄素依赖Diels-Alder[4+2]环加成酶的结构和催化机制方面取得新进展
Diels-Alder(D-A)反应是人们所最为熟知的有机人名反应之一,并被广泛地应用于合成化学、药物化学、材料化学和化学生物学的研究中。人们基于路易斯酸活化以及氢键活化策略设计了不同的小分子催化剂以催化D-A反应,同时还通过分子定向进化的方法筛选得到了能够催化D-A反应的RNA酶和DNA酶。但是长期以来,人们对于自然界中是否存在天然的能够催化D-A反应的酶这一问题以及其可能的催化机制,却一直存在争议。
在前期的研究工作中,中科院上海有机所生命有机国家重点实验室刘文课题组在国际上发现了吡咯吲哚霉素(pyrroindomycin)的生物合成途径中涉及到了两步绝对酶依赖的D-A反应,分别由PyrE3和PyrI4这两个酶催化完成(J. Am. Chem. Soc., 2012, 134, 17342-17345;Nat. Chem. Biol., 2015, 11, 259-265);与中科院上海有机化学研究所生命有机国家重点实验室潘李峰课题组合作,报道了首个非辅因子依赖的D-A酶PyrI4及其与小分子产物的复合物晶体结构(Cell Chem. Biol., 2016, 23, 352-360)。基于前期的研究成果,刘文课题组提出了天然D-A酶催化过程中的“过渡态模板作用”;为了实现这一作用,酶与小分子底物相互作用过程中会放出大量能量并发生三维构象的变化,从而提供一个用于稳定D-A反应过渡态的完美反应微环境(Curr. Opin. Chem. Biol., 2016, 31, 95-102)。
在本研究中刘文课题组与潘李峰课题组再次合作,解析了黄素FAD依赖的D-A酶PyrE3的晶体结构,这也是国际上首个结构被解析的黄素依赖D-A酶(Cell Chem. Biol., 2018, DOI: 10.1016/j.chembiol.2018.03.007)。基于所获得的晶体结构,他们综合使用分子对接模拟、生物信息学分析、氨基酸定点突变、光谱学分析等手段指出了PyrE3的催化空腔和FAD在催化过程中的作用。他们发现,与其他FAD依赖的加氧酶不同,PyrE3在催化反应的过程中并不要求FAD发生氧化态的变化;FAD能够稳定蛋白的结构,并通过与底物分子形成氢键来释放结合能并稳定其过渡态构象。国际上先后有几例辅因子(如腺苷甲硫氨酸等)依赖的D-A酶报道,但是这些辅因子在D-A酶催化过程中的作用并不明确。本研究的相关成果为今后人工设计、改造或开发协同反应的生物大分子催化剂(尤其是辅因子依赖的蛋白)提供了重要的实验基础和理论方向,可能在未来的合成化学和合成生物学研究中发挥重要的作用。
刘文课题组的博士生郑庆飞(已毕业)和潘李锋课题组的博士生龚余康为本文的共同第一作者。上述研究工作得到了科技部、国家自然科学基金委、上海市科委、中国科学院战略性先导科技专项(B类)以及生命有机化学国家重点实验室的大力资助。
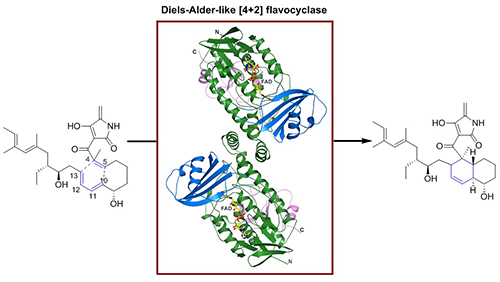
黄素依赖蛋白Pyr3催化的DA-like[4+2]环加成反应
附件下载: